
Ce que ça apporte (le nouveau parseur + le middleware)
Jusqu’ici, le parseur NCBI intégré à ezPAARSE permettait principalement d’identifier le rtype (ARTICLE, SEARCH, ABS) et le type mime (HTML ou PDF).Avec la nouvelle configuration (parseur+middleware) que nous vous proposons, l’identification est beaucoup plus complète et peut se poursuivre via unpaywall et crossref (voir la section détails). Cela a cependant un coût, qui sur notre échantillon triple les temps de traitement et ajoute un volume de données d’usage d’un peu moins de 20%. Mais cela dépend bien sûr du type de consultation de vos utilisateurs, en effet, le cache d’ezPAARSE peut entrer en jeu et il est assez efficace.
PubMed Central® (PMC) est une archive gratuite en texte intégral de la littérature biomédicale et des sciences de la vie et fait partie de la plateforme NCBI. Il existait déjà un parseur dans ezPAARSE depuis 2 ans. Il a été mis à jour récemment, pour étendre ses capacités de reconnaissance, par un contributrice américain Kgilje (déjà active sur d’autres parseurs), que nous remercions ici.
Comment configurer ezPAARSE pour utiliser ces nouvelles fonctionnalités de reconnaissance ?
Ce nouveau middleware est maintenant intégré dans ezPAARSE, et vous pouvez l’activer lors de vos prochains traitements de log. Par défaut le middleware est bien configuré (usage FAIR de l’API de NCBI). Attention à bien respecter les usages si vous en changez la configuration.
Via l’interface
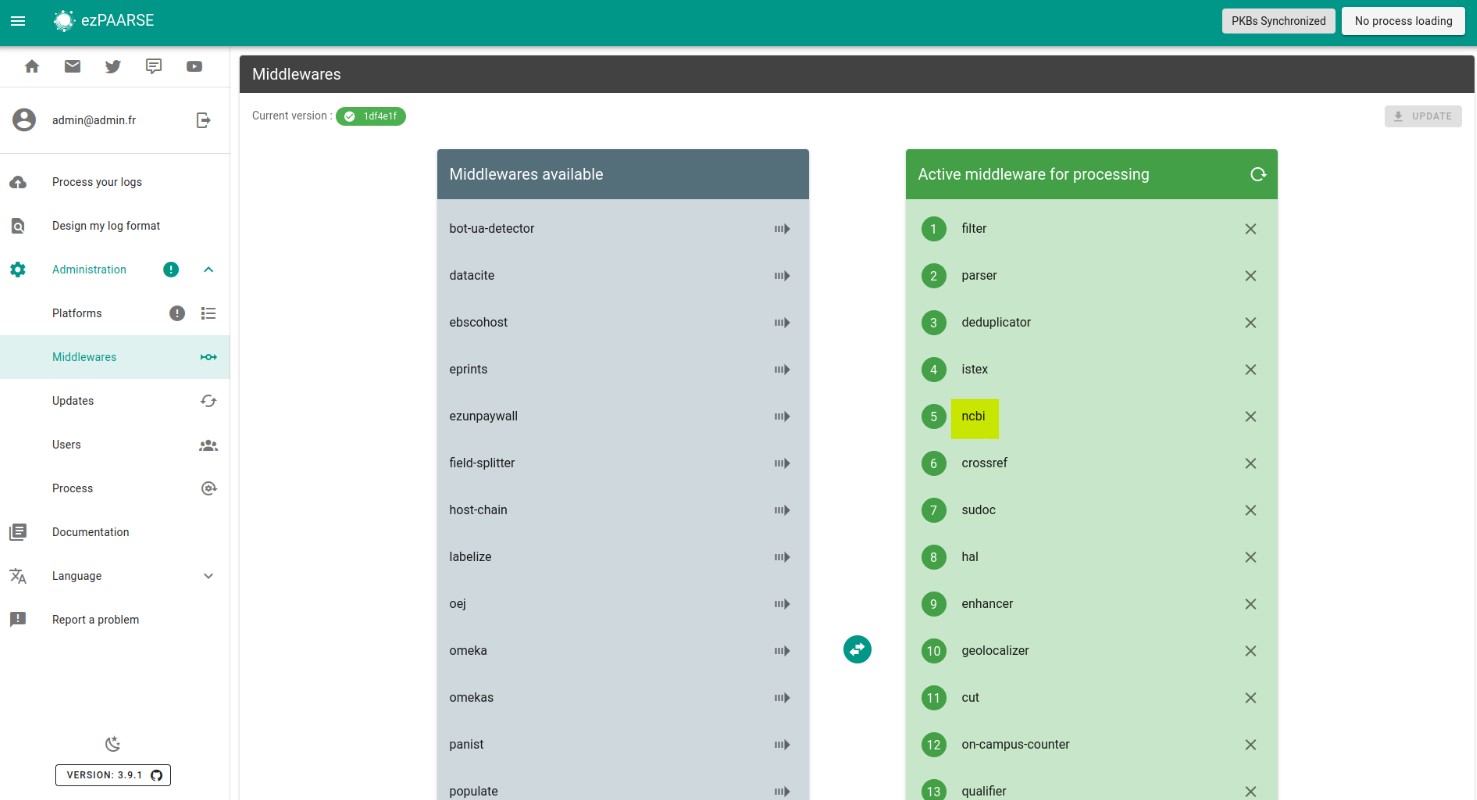
Pour utiliser le middleware NCBI, vous devez être administrateur (ou correspondant technique) d’un ezPAARSE installé dans votre établissement (correspondant technique) et vous rendre dans la partie middleware de l’interface. Il faut ensuite placer le middleware ncbi avant crossref (système de glisser-déposer sur l’interface).
En ligne de commande dans vos traitements quotidiens
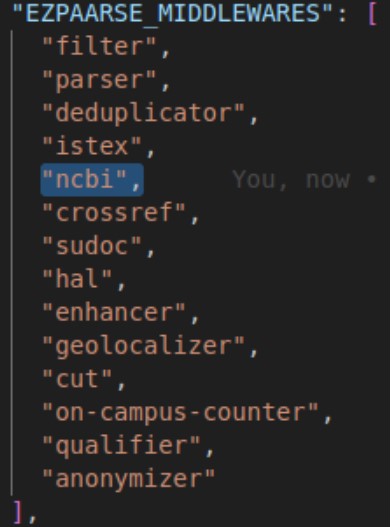
Pour utiliser le middleware systématiquement dans tous vos traitements, vous pouvez ajouter le middleware dans votre fichier config.local.json dans la clé EZPAARSE_MIDDLEWARES. Pour profiter pleinement de l’enrichissement, il est conseillé de le placer juste avant crossref.
Pour intégrer l’enrichissement ncbi occasionnellement dans vos traitements batch, il faut ajouter le paramètre à votre ligne de commande :

Mais en détail, comment cela se passe-t-il ?
Le parseur NCBI dans ezPAARSE dispose d’une analyse sur AnalogIST qui permet de comprendre les types d’accès aux données de la plateforme :
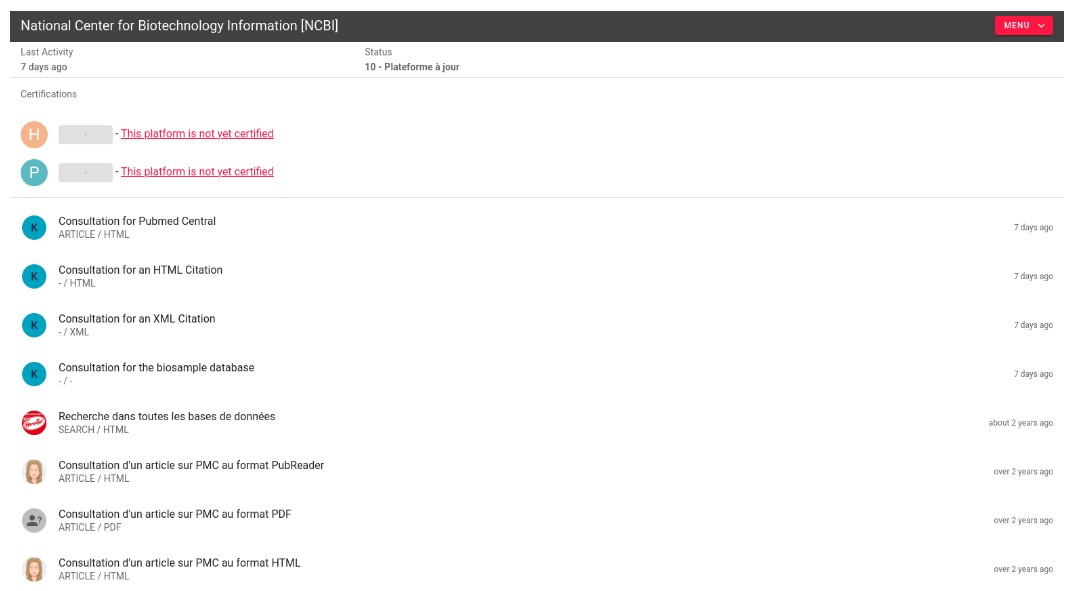
http://analyses.ezpaarse.org/platforms/542571dbc703a13f0fccfa3c
L’accès aux documents en texte intégral (ou fulltext) mis en ligne avec un identifiant de type PMCXXXX présent dans l’URL de consultation.
Exemple d’URL : /pmc/articles/PMC3555666 (PUB MED CENTRAL)
NCBI met à disposition une API qu’on interroge avec cet identifiant PMC et qui nous permet de récupérer des informations bibliographiques précises sur le document qui a été consulté. Pour que cet enrichissement avec des métadonnées issues de l’API NCBI puisse avoir lieu dans ezPAARSE lors des traitements de logs, il aura fallu créer un middleware : un petit programme qui est déclenché au cours du traitement ezpaarse, selon le même principe qu’avec le middleware Crossref.
Les métadonnées récupérées depuis l’API NCBI sont :
- print_identifier (ISSN)
- online_indentifier (E-ISSN)
- publication_title
- doi
- title
Voici la proposition du middleware (image du dépôt dans github)
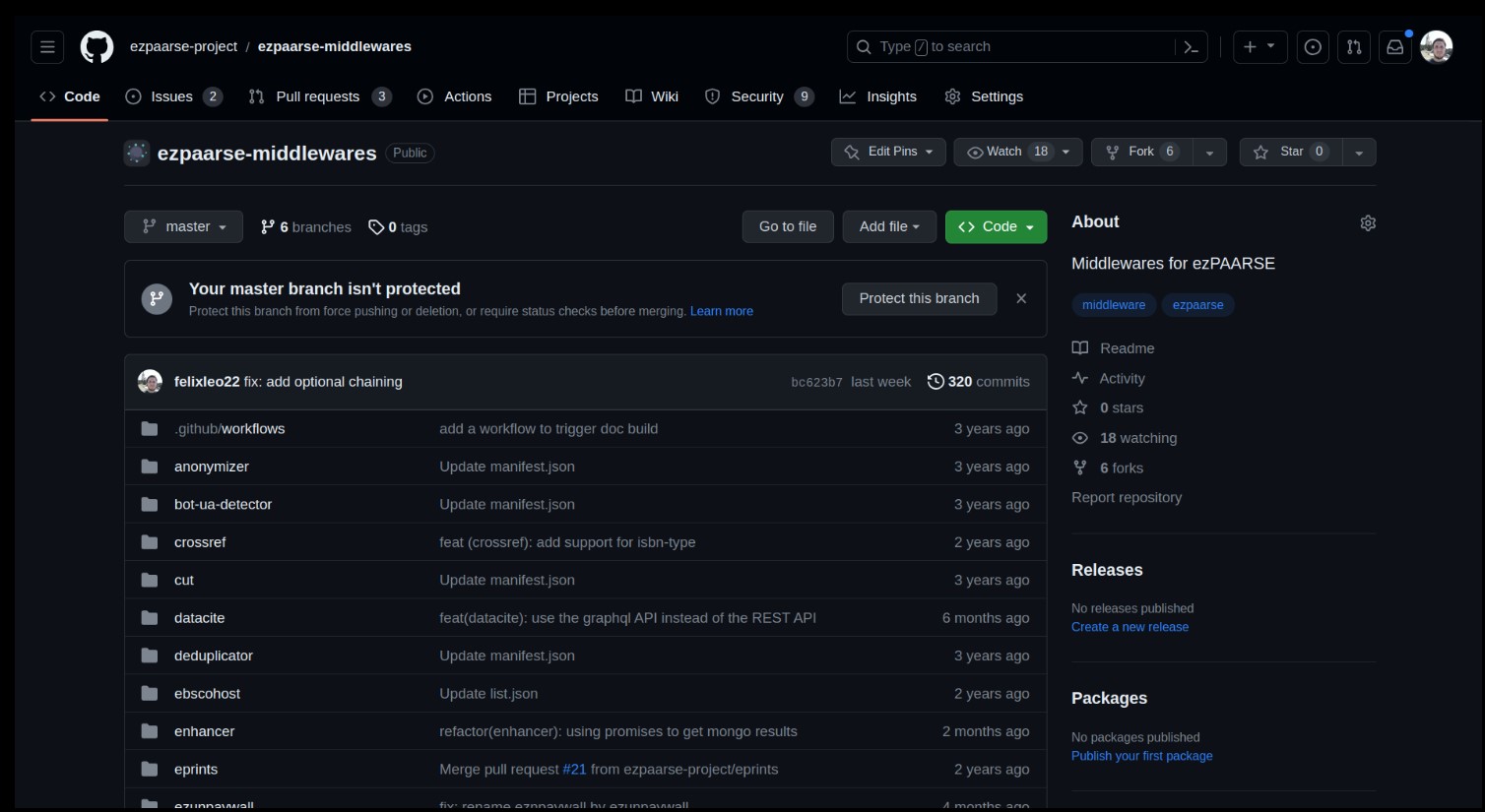
L’équipe ezTEAM a validé la pull request récente sur Github.
Le parseur de NCBI a également été mis à jour.
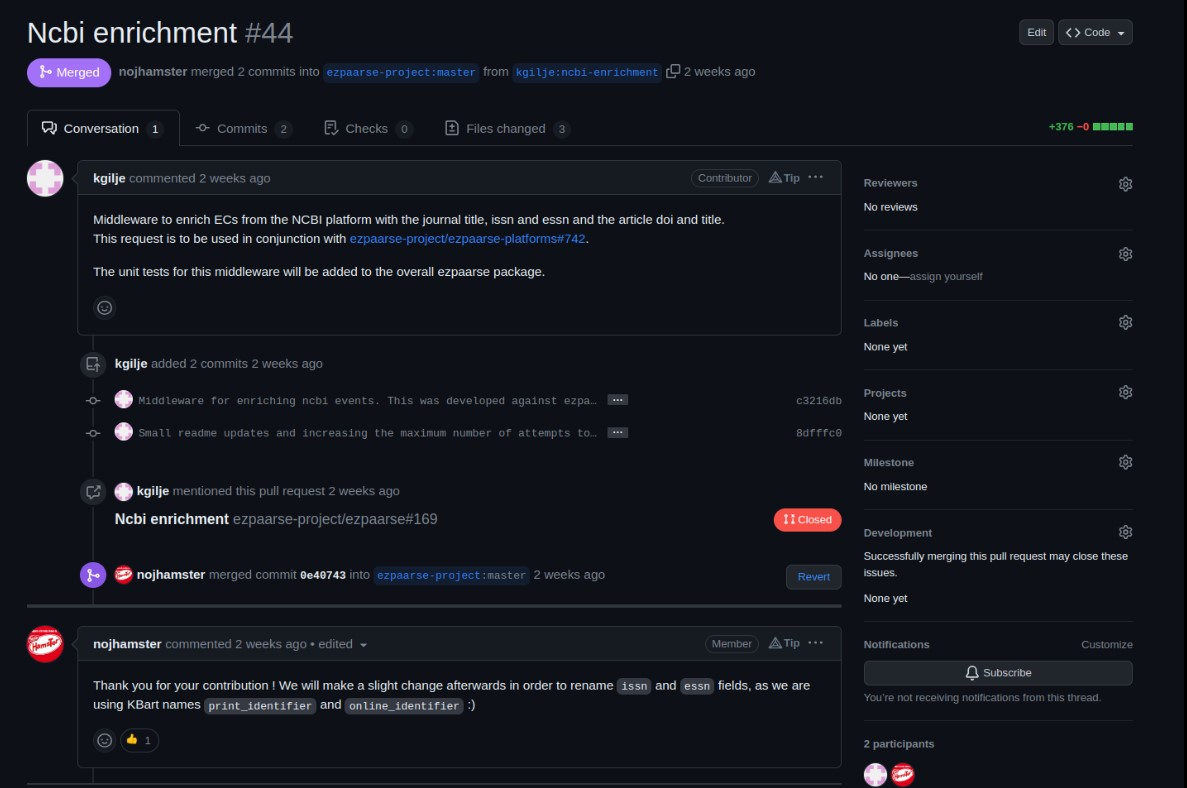
Nous avons testé le middleware NCBI développé. Voici le résultat (image d’un fichier d’EC avec les champs récupérés) :
Avant, sans le middleware NCBI
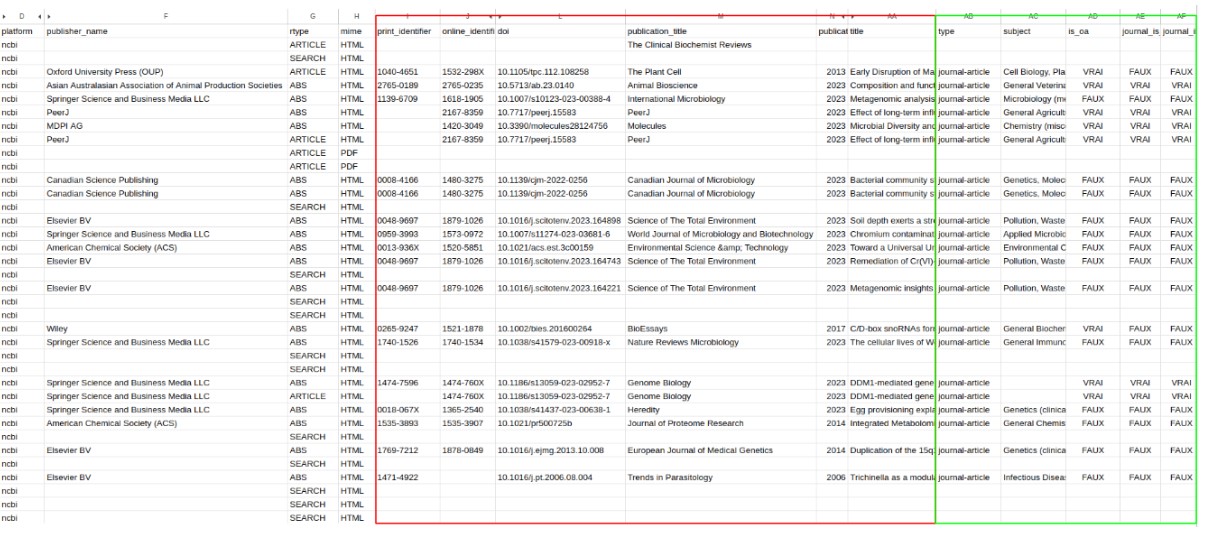
Ensuite, avec le middleware NCBI
Ce qui est entouré en rouge est l’enrichissement apporté par le middleware NCBI avec en vert l’enrichissement crossref et unpaywall.
Cet enrichissement est possible grâce au DOI rapatrié par le middleware NCBI.
Conclusion
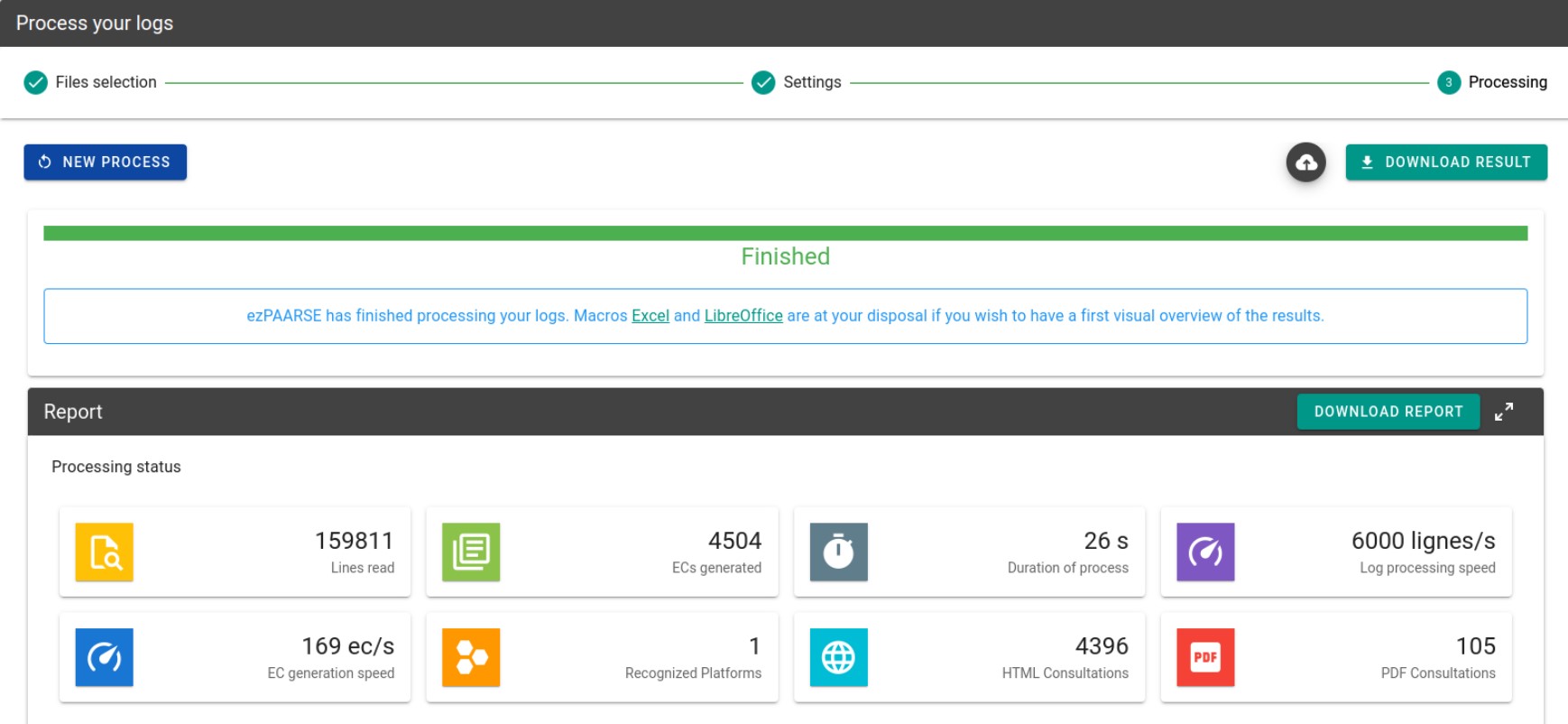
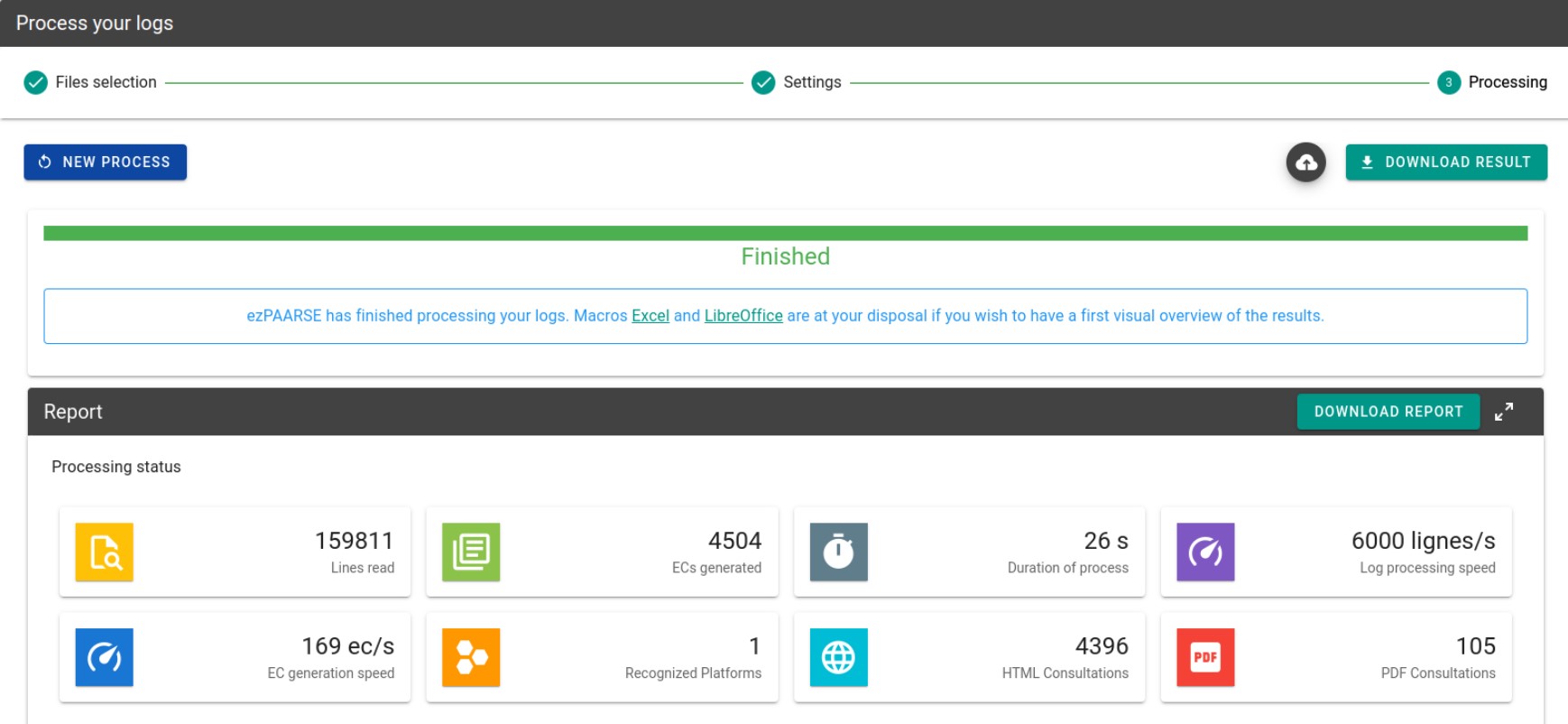
L’ajout d’un middleware effectuant des requêtes d’enrichissement sur un site externe peut avoir des conséquences sur les temps de traitement. Nous avons procédé à une évaluation de ces différences visibles dans les copies d’écrans ci-dessous. Le coût est assez élevé (selon les consultations de vos utilisateurs), puisque dans l’exemple de notre échantillon, il provoque un triplement du temps de traitement, ainsi qu’un volume d’EC produits augmenté de 18%, mais ce surcoût est le prix d’une qualité bien meilleure des enrichissements.
Sans le middleware NCBI
Avec le middleware NCBI
L’ augmentation du temps vient du fait qu’on interroge une API et qu’aussi, lorsqu’on récupère un DOI, et qu’on interroge crossref (ce qui peut être long) et unpaywall
Si vous avez besoin de renseignements complémentaires, vous pouvez nous contacter à ezteam@couperin.org.
Bonne journée.
Léo Felix et Frédéric Truong pour ezTEAM